トップページ > 研究組織一覧 > 分野・独立ユニットグループ > がん治療学研究分野 > 研究成果の概要 > データ駆動型アプローチ:大規模データベースと独自細胞株パネルを統合した合成致死標的の探索
データ駆動型アプローチ:大規模データベースと独自細胞株パネルを統合した合成致死標的の探索
1. 概要:なぜ「データ解析」が必要なのか?
がん細胞には、数千~数万の遺伝子変異が存在します。その中から「どの遺伝子を阻害すればがんが死ぬか(合成致死)」を実験室の総当たり戦で探すには、膨大な時間とコストがかかります。 私たちは、世界中の細胞株の生存必須性データを集約した公共データベース(DepMap)と、当研究所が独自に構築した希少がん・日本人特有のがん細胞株パネルのデータを統合解析することで、実験室での検証前に「有望な標的」をピンポイントで予測するシステムを構築しています。
この「In silico(コンピュータ上)での予測」と「In vitro / In vivo(実験室)での検証」を高速に回すことで、従来の手法では見落とされてきた治療標的の同定に成功しています。
2. 独自の探索アルゴリズム(Methodology)
- 独自の探索アルゴリズム(Methodology)
私たちは、Broad Instituteが公開しているCancer Dependency Map (DepMap) などの大規模データセットを活用し、特定のがん(遺伝子欠損がん)において「特異的に依存度が高い遺伝子(弱点)」を抽出する独自の解析パイプラインを確立しました。
【解析のステップ】
- Dependency Scoreの算出: 1000種類以上のがん細胞株における、全遺伝子(約17,000遺伝子)のCRISPR/RNAiスクリーニングデータを取得します。
- 「引き算」による標的抽出: 特定の遺伝子(例:ARID1A)が「欠損しているがん細胞群」と「正常ながん細胞群」を比較し、欠損群でのみ生存に必須となる(Dependency Indexが低い)遺伝子を探索します。
- 組織特異性の考慮: 単に全がん種で解析するのではなく、「卵巣がん」「肉腫」など組織型を限定して解析することで、そのがん種特有の弱点(コンテキスト依存的な合成致死)を見逃さずに捉えます。
3. 成功事例:ARID1A欠損がんに対する「USP8」の同定
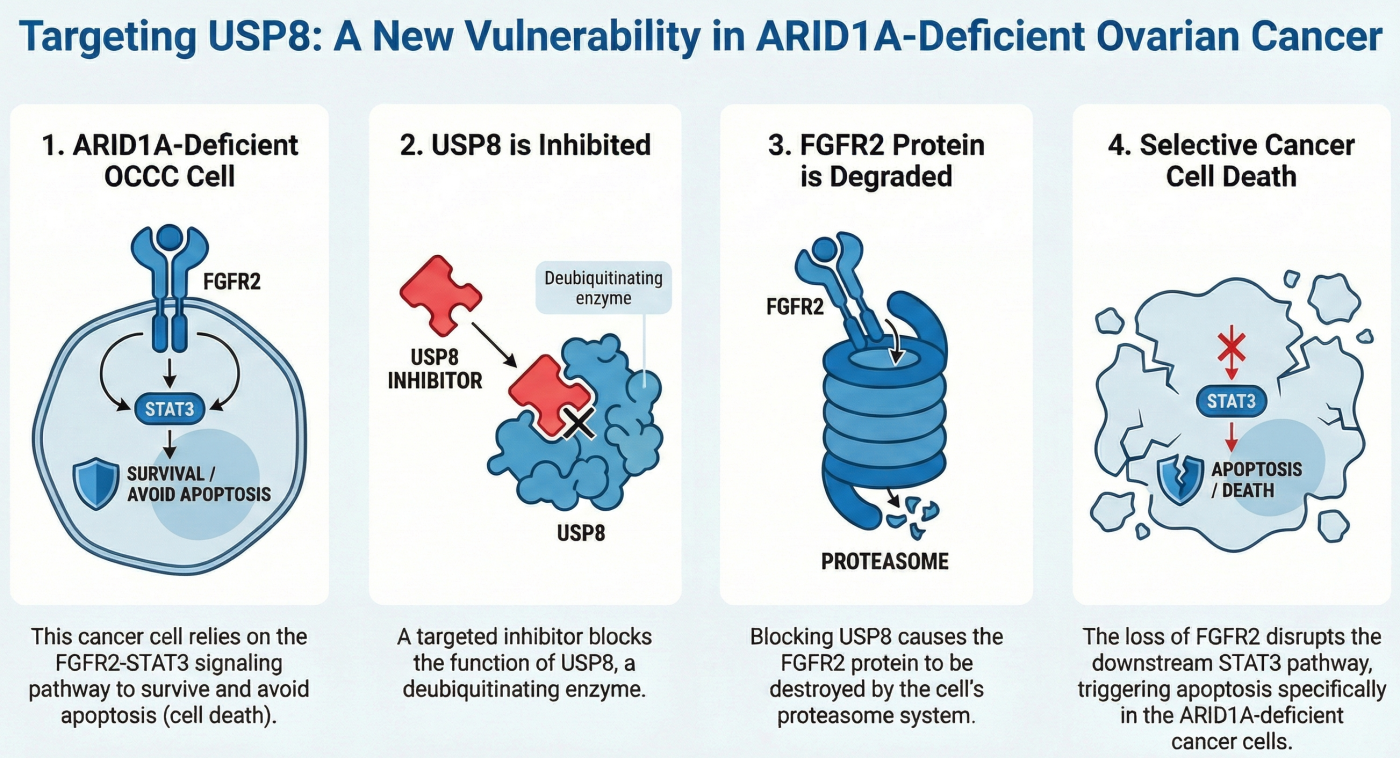
このデータ駆動型アプローチにより、私たちは難治性である卵巣明細胞がん(OCCC)の50%以上で欠損が見られるARID1A遺伝子に対する新しい合成致死標的として、脱ユビキチン化酵素USP8を同定しました。
発見の経緯
DepMapデータベースを用い、卵巣がん細胞株に限定して「ARID1A欠損細胞が特異的に依存する遺伝子」を解析した結果、USP8がトップランクの候補として抽出されました。実際に実験室で検証したところ、USP8を阻害するとARID1A欠損細胞のみが特異的に死滅することが確認されました。
解明されたメカニズム:FGFR2の分解誘導
なぜUSP8阻害が効くのか? そのメカニズム解析においても、網羅的なユビキチン化プロファイリングなどの最新技術を駆使しています。
- USP8の役割: 通常、USP8はFGFR2(線維芽細胞増殖因子受容体)のユビキチンを取り除くことで、FGFR2が分解されるのを防いでいます。
- 阻害時の作用: USP8を阻害すると、ARID1A欠損細胞ではFGFR2が過剰にユビキチン化され、プロテアソームによって分解されてしまいます。
- 細胞死の誘導: FGFR2が消失することで、下流の生存シグナルであるSTAT3の活性が低下し、アポトーシス(細胞死)が誘導されます。
- 掲載論文: npj Precision Oncology. 2025
4. 今後の展望:希少がん・難治性がんへの応用
USP8の事例は、データ解析から始まった研究が、新しい分子メカニズムの解明、そしてマウスモデルでの治療効果の実証(前臨床試験)まで繋がった好例です。 現在、私たちはこのアプローチをさらに拡大し、以下のがん種・遺伝子変異に対する解析を進めています。
- 対象: スキルス胃がん、膵臓がん、小児・AYA世代の肉腫
- 戦略: 公共データベースにない「日本人特有の希少がん細胞株」のデータを独自に追加し、AI(機械学習)を用いた予測精度の向上にも取り組んでいます。

